Coordinador
- Javier Paz-Ares, Centro Nacional de Biotecnología, C.S.I.C., Madrid.
Investigador principal del subproyecto
- José Luis Micol, Instituto de Bioingeniería, Universidad Miguel Hernández.
Otros investigadores del subproyecto
- María Rosa Ponce, Instituto de Bioingeniería, Universidad Miguel Hernández.
Proyecto integrado de genómica funcional en Arabidopsis (GEFA)
Casi todas las parcelas de la Biología del siglo XXI están sufriendo una profunda transformación causada por la rápida generalización de los métodos de la Genómica, el análisis a gran escala de la estructura y la función de los genes. Esta renovación metodológica y conceptual es sólo comparable a la que supuso la revolución industrial de finales del siglo XVIII, que introdujo los sistemas de producción mecanizados que determinaron la sustitución de la manufactura manual y doméstica por la producción fabril a gran escala, e implica grandes cambios en los enfoques experimentales, en la naturaleza de las herramientas instrumentales y en la magnitud de las tareas investigadoras que deben acometerse. Estos cambios no pueden darse de manera efectiva sin profundas modificaciones en la mentalidad de los investigadores y en el comportamiento de los colectivos que comparten objetivos científicos. Conscientes de ello, los grupos de investigación que suscriben esta propuesta pretenden implantar nuevas estructuras, recursos y servicios nacionales que posibiliten la aplicación de enfoques genómicos a la investigación básica y aplicada en la biología de las plantas. Para ello, se propone poner en marcha varias iniciativas de alto rendimiento y a gran escala, empleando como organismo experimental a Arabidopsis thaliana, el sistema modelo por excelencia en biología vegetal. En el estudio de esta especie concentran sus esfuerzos varios grupos de élite españoles, cuyo dinamismo y productividad demostrada les convierten en un colectivo ideal para ensayar un modelo de organización de la actividad científica sin precedentes en nuestro país, que puede posteriormente aplicarse a otros colectivos científicos españoles. Concretamente, proponemos actividades en siete frentes, todas ellas plenamente congruentes con las tendencias más recientemente acusadas a nivel internacional en el marco de programas de genómica extranjeros, a los que complementan y refuerzan. Estas actividades son las siguientes: 1) Análisis del transcriptoma, mediante la producción y utilización de “chips” de DNA; 2) Análisis del fosfoproteoma y el proteoma nuclear; 3) Obtención de mutantes nulos mediante mutagénesis de inserción, llevando a cabo la secuenciación de las secuencias genómicas que flanquean a cada inserción; 4) Obtención de series alélicas de mutaciones puntuales y deleciones en genes específicos; 5) Cartografía génica automatizada, para facilitar la clonación posicional de genes a partir de sus alelos mutantes no señalizados; 6) Identificación y clonación de todas las pautas de lectura abiertas del cromosoma 4; y 7) Explotación de la variabilidad natural, mediante la recolección y caracterización de estirpes silvestres en España y la realización de estudios filogenómicos para la identificación de regiones reguladoras de la expresión génica. Estas siete iniciativas supondrán la puesta en marcha de siete servicios de cuya actividad serán beneficiarios todos los grupos de investigación que suscriben esta propuesta.
Subproyecto: Cartografía génica automatizada
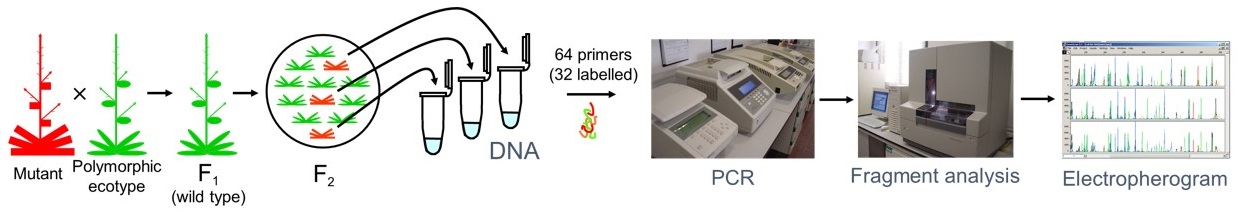
El objetivo primordial de las aproximaciones genéticas al estudio de la función de un gen es la identificación de éste a partir del aislamiento de mutantes. Una estrategia comúnmente utilizada en Arabidopsis thaliana es la obtención de mutantes señalizados mediante mutagénesis insercional, lo que en ocasiones permite la identificación casi inmediata de las secuencias que flanquean a la inserción del mutágeno, que corresponden al gen dañado por la mutación. Sin embargo, son numerosos los inconvenientes del uso del mutágeno insercional más eficaz en Arabidopsis thaliana, el ADN-T del plásmido Ti de la bacteria Agrobacterium tumefaciens, ya que genera en muchos casos inserciones múltiples e independientes, en otros causa mutaciones no señalizadas debidas a una inserción abortiva, y forma habitualmente concatémeros de gran tamaño e internamente reorganizados. Estas peculiaridades de los vectores derivados del ADN-T impiden en muchos casos la clonación de los genes dañados por sus inserciones. A todo lo anterior debe añadirse que la práctica totalidad de las mutaciones causadas por el ADN-T son nulas y frecuentemente causan letalidad recesiva.
Los mutágenos físicos y químicos constituyen una alternativa a la mutagénesis insercional. La mayor de las objeciones que se han opuesto a su uso consiste en que la clonación de los genes afectados por estos mutágenos debe llevarse a cabo mediante aproximaciones posicionales, que han sido consideradas durante mucho tiempo lentas y tediosas. Este punto de vista, sin embargo, está cambiando merced a la disponibilidad de la secuencia completa del genoma de Arabidopsis thaliana, el incremento de la densidad de los marcadores moleculares en el mapa genético de esta planta, y el desarrollo de técnicas de cartografía génica de gran rendimiento y alta resolución. Por otra parte, la mutagenicidad de los mutágenos físicos y químicos es muy superior a la del ADN-T, pudiendo inducir tanto alelos nulos letales como hipomorfos viables.
El objetivo específico de este subproyecto es la creación y puesta en marcha de un servicio de cartografía génica, automatizada y de alta resolución, de mutantes de Arabidopsis thaliana no señalizados. Este servicio trabajará con muestras remitidas por los grupos de investigación que suscriben esta propuesta, consistentes en 50 individuos de la F2 de un cruzamiento entre una planta homocigótica para una mutación de interés y un ecotipo distinto de su ancestro silvestre. Su ADN será extraido por el servicio de cartografía, para llevar a cabo un análisis del ligamiento a SSLP, que permitirá determinar la posición de mapa de la mutación a estudio dentro de un intervalo de 15 cM. La cartografía de alta resolución se realizará en una segunda etapa, en la que se emplearán 450 plantas F2 adicionales para identificar, mediante análisis del ligamiento a SSLP, un cóntigo de dos o tres clones BAC correspondiente al segmento del genoma de Arabidopsis thaliana en el que radica el gen afectado por la mutación de interés.